Back to top
miCORE Resequencing

- vererbte oder erworbene Mutationen innerhalb eines Genoms aufzuspüren.
- genomweite Vergleiche durchzuführen.
- Stamm oder Zelltyp auf genomischer Ebene zu verifizieren.
- Off-Target-Mutationen in Ihrem CRISPR/Cas9-Editierexperiment nachzuweisen.
Übersicht
Überlegungen vor Beginn eines whole genome sequencing Projekts:
- Wissenschaftliches Ziel
- Qualität des Referenzgenoms
- SNV Nachweis (Variant Calling)
- Optimale Abdeckung / Sequenziertiefe & Leselänge
- Besteht der Verdacht auf signifikante DNA-Kontaminationen?
Lassen Sie sich von uns beraten - vom Design bis zur Analyse
Beispiele für Projekte, bei denen die Whole Genome Sequenzierung zum Einsatz kommt:
- Nachweis von Mutationen, die von Krebszellen erworben wurden - von SNPs bis zu großen strukturellen Veränderungen.
- Nachweis von Insertionsstellen
- Mutationsverifizierung, Ausschluss von Off-Target-Mutationen
- Genetische Veränderungen in Zuchtstudien
Anwendungen im Zusammenhang mit der Whole Genome Sequenzierung:
- RNA Sequencing
- Whole Exome Resequencing
- De Novo Sequencing
Workflow

Resultate
Ohne Bioinformatik
Rohdaten:
Wenn kein Analysemodul bestellt wird, liefert Microsynth für Whole Genome Sequencing die folgenden Hauptergebnisse:
- Bewertung der Quantität und Qualität der Sequenzierung (im .xlsx-Format)
Bewertung der Quantität und Qualität der Sequenzierdaten. - Rohdaten (pro Probe, im .fastq-Format)
Mit den Rohdaten können Sie Ihre eigenen Analysen durchführen oder jedes sequenzierte Nukleotid zurückverfolgen. - Zusammenfassender Projektbericht (.pdf-Format)
Der Bericht enthält eine Zusammenfassung der wichtigsten Parameter des Projekts.
Mit Bioinformatik
Bioinformatische Standardanalyse:
Für unsere Resequenzierungsanwendungen bietet das Analysemodul von Microsynth zusätzlich zu den Rohdaten eine Vielzahl von Einblicken, um Ihre wissenschaftlichen Ziele zu erreichen:
- Bewertung der Quantität und Qualität der Sequenzierung (im .xlsx und .html Format)
Eine umfassende Auswertung der Quantität und Qualität der Sequenzierung, die wichtige Erkenntnisse liefert. - Alignment/Map-Dateien und Indizes (in den Formaten .bam und .bai)
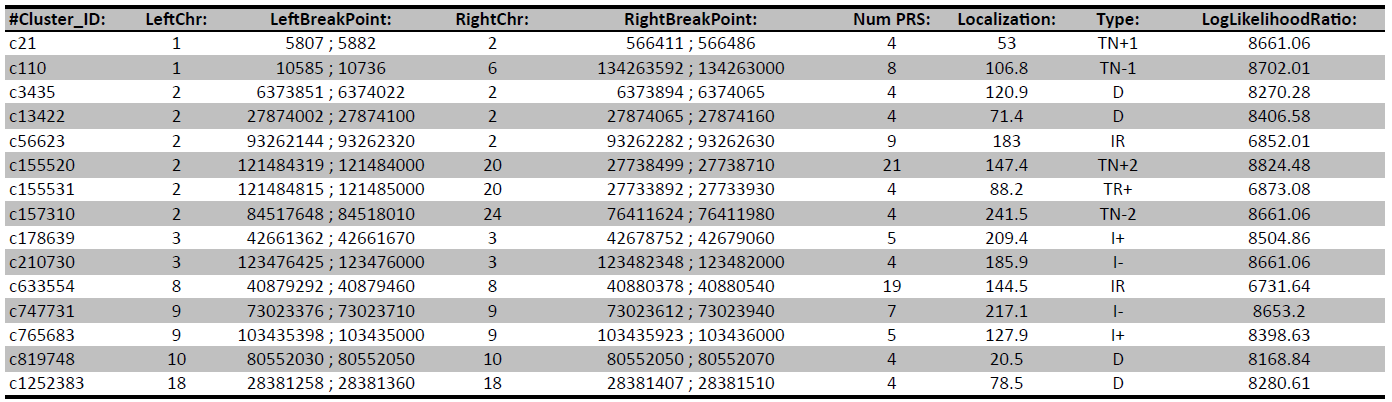
Zugriff auf Alignment- und Map-Dateien mit den zugehörigen Indizes. - Analyse der genomischen Abdeckung und Lesetiefe (in den Formaten .tsv und .bed; siehe Abbildung 1)
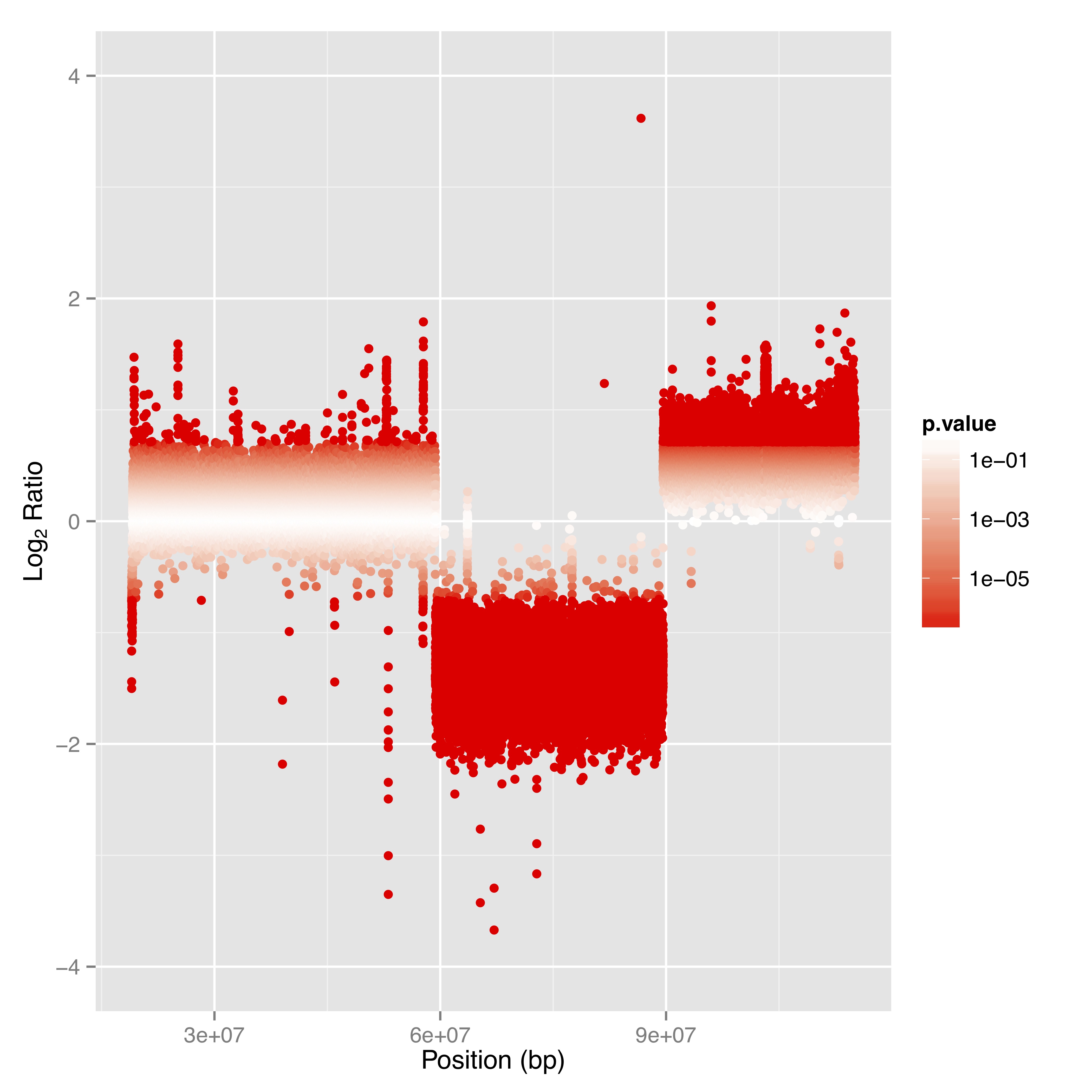
Erhalten Sie Einblicke in die Genomabdeckung und Lesetiefe anhand der Ergebnisse, die in den Formaten .tsv und .bed bereitgestellt werden. - Strukturelle Variationen & Kopienzahlanalyse (siehe Tabelle 1 & Abbildung 2)
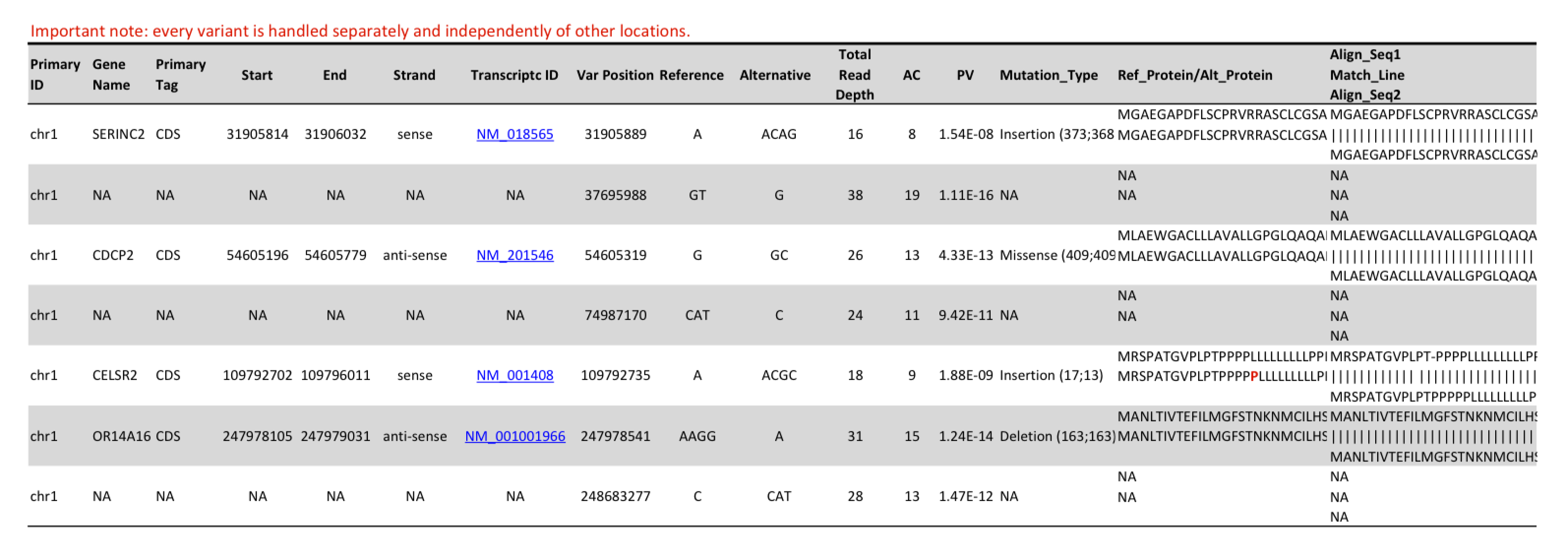
- Variant Calling von SNVs und kleinen InDels (im .vcf-Format, siehe Tabelle 2 und Tabelle 3 )
Identifizierung von Einzelnukleotid-Varianten (SNVs) und kleinen Insertionen/Deletionen (InDels < 50 bp) mit den Ergebnissen der Variantenidentifizierung im .vcf-Format. - Annotation von Variationen mit Konsequenzen auf Aminosäureebene (im .html-Format, siehe Tabelle 4)
Verständnis der Variationen mit potenziellen Konsequenzen auf Aminosäureebene, dargestellt im .html-Format (verfügbar für die meisten Modellorganismen / wenn das Referenzgenom entsprechend annotiert ist).
Optional:
- Filterung von Varianten gegen ein Hintergrundmuster (im .html-Format)
Passen Sie Ihre Analyse an, indem Sie Varianten gegen eine Hintergrundprobe filtern und die Ergebnisse im .html-Format erhalten. - Probenkonsensussequenz (im .fasta- und .gb-Format)
Erhalten Sie eine Probenkonsensussequenz, die zu einer chimären Sequenz führen könnte, im .fasta- und .gb-Format.
Zusätzliche bioinformatische Analyse (kostenpflichtig)
Genomische Epidemiologie (nur bei Prokaryonten):
- Screening auf Übereinstimmungen mit Resistenz- und Virulenzdatenbanken und Mykotoxin-Genen (im .tsv-Format)
Identifizierung von Übereinstimmungen mit Resistenz- und Virulenzdatenbanken mit Bereitstellung der Screening-Ergebnisse im .tsv-Format. - Phylogenetische Typisierung von Probenstämmen (im .pdf-Format)
Verstehen Sie die phylogenetische Verwandtschaft Ihres Probenstamms anhand eines übersichtlichen .pdf-Berichts.
Diese Ergebnisse geben Ihnen einen umfassenden Überblick über Ihre Re-Sequenzierungsanalyse und ermöglichen es Ihnen, aussagekräftige Erkenntnisse zu gewinnen und fundierte Entscheidungen zu treffen.
Tabelle 1: Dieser Auszug aus einer Ergebnistabelle zeigt die detektierten Einzelnukleotidvariationen, kleine Insertionen und Deletionen und deren Annotation.
Bearbeitungszeiten
- Lieferung der Daten innerhalb von 20 Arbeitstagen nach Probeneingang (einschließlich Vorbereitung der Bibliothek und Sequenzierung).
- Weitere 3 bzw. 6 Arbeitstage für Datenanalyse (Bioinformatik; kleine bzw. große Genome).
- Express-Service auf Anfrage.