Back to top
ChIP Sequenzierung

Verwenden Sie ChIP-Sequenzierung, um:
- die genomweite Transkriptionsregulation in einer vergleichenden Umgebung zu untersuchen
- den aktiven Teil des Chromatins zu untersuchen
Übersicht
Überlegungen vor dem Start eines ChIP-Sequenzierungsprojekts:
- Gibt es ein geeignetes Referenzgenom?
- Punktuelle oder breite Quelle?
- Sequenziertiefe (Sensitivität)?
- Experimenteller Aufbau (Replikate und Bedingungen, Eingangskontrolle)?
Lassen Sie sich von uns beraten - vom Entwurf bis zur Analyse
Beispielprojekte mit ChIP-Sequenzierung:
- Entdeckung von neuen Transkriptionsfaktor-Bindungsstellen
- Untersuchung von Histon-Modifikationen
- Selektive Nutzung von Transkriptionsfaktoren
- Neuartige Aktivierung von Signalwegen durch bekannte Transkriptionsfaktoren
Anwendungen im Zusammenhang mit der ChIP-Sequenzierung:
- mRNA sequencing
- small and miRNA sequencing
Workflow
Ein typischer Workflow für ein ChIP-Sequenzierungsprojekt ist in der folgenden Grafik dargestellt. Bitte beachten Sie, dass unsere hochmodularen Prozesse Ihnen verschiedene Einstiegs- und Ausstiegsmöglichkeiten bieten. Ob Sie Ihr gesamtes NGS-Projekt an Microsynth auslagern oder nur Teile davon, bleibt Ihnen überlassen.
Resultate
Die von unserem Analysemodul erzeugten Ergebnisse helfen bei der Beantwortung von drei Hauptfragen eines ChIP-Sequenzierungsexperiments, die zum Beispiel darauf abzielen, neue Muster der Genregulation zu finden.
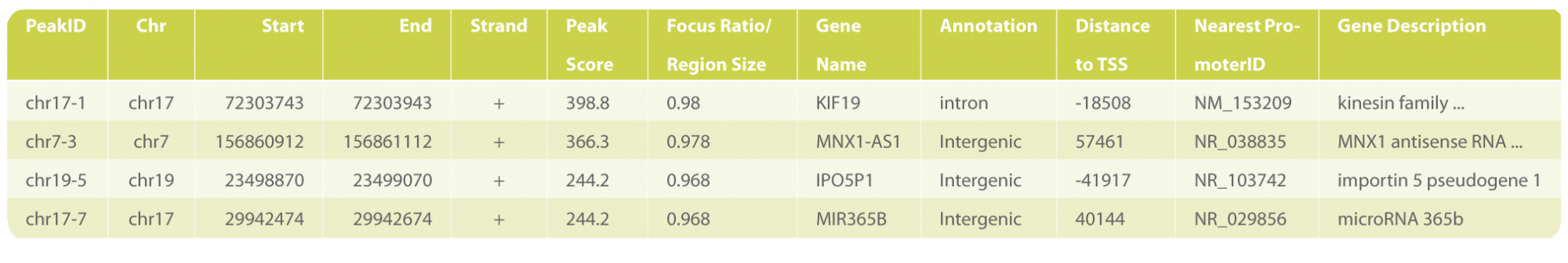
- Wo auf dem Referenzgenom befinden sich signifikante Peaks? (siehe Tabelle 1)
- Welches sind die gemeinsamen Motive und sind diese neuartig? (siehe Abbildung 1)
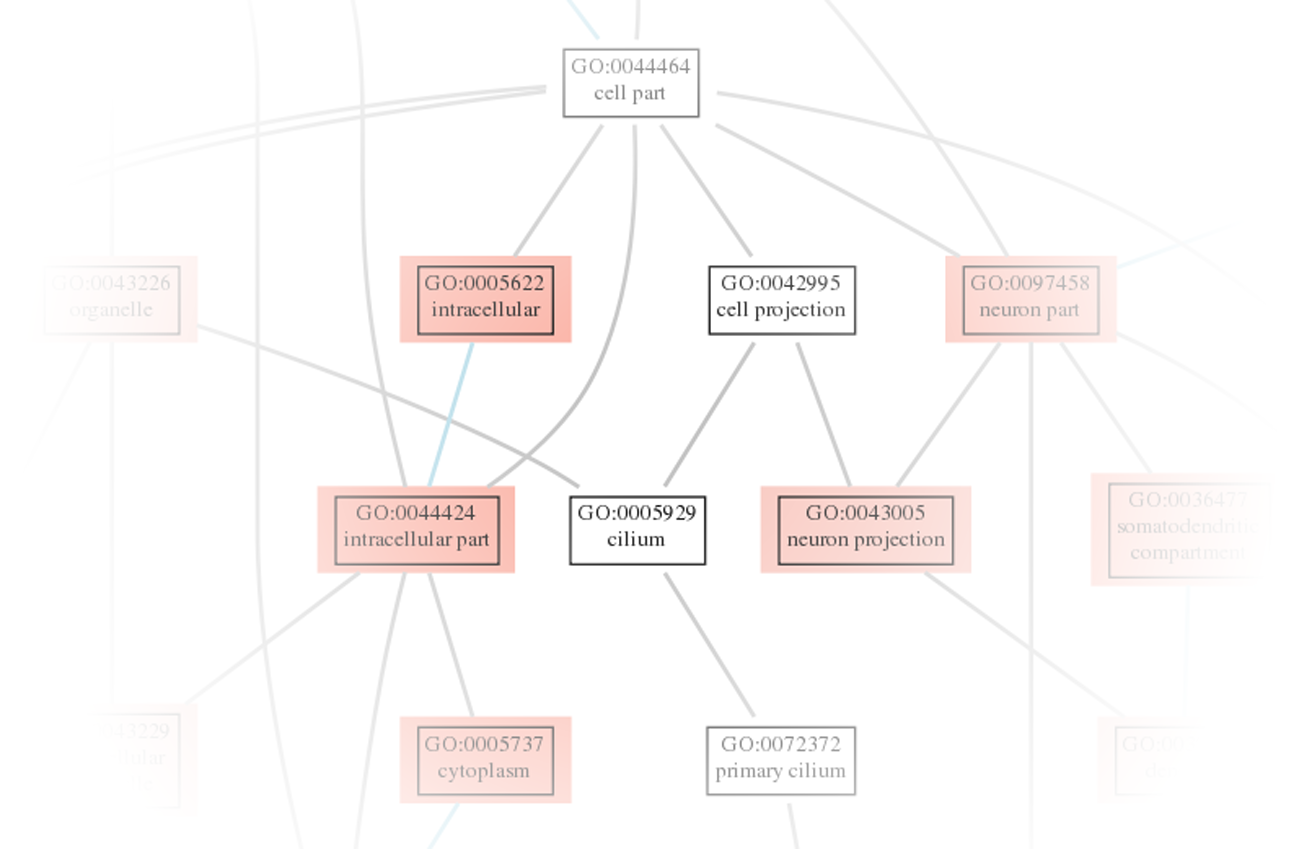
- Welche Signalwege können durch die erfassten regulatorischen Elemente beeinflusst werden? (siehe Abbildung 2)
Bearbeitungszeiten
- Lieferung der Daten innerhalb von 25 Arbeitstagen nach Probeneingang (inklusive Library Erstellung und Sequenzierung)
- Weitere 10 Arbeitstage für die Datenanalyse (Bioinformatik)
- Express-Service auf Anfrage möglich