
Back to top
siRNA für Gen-Silencing

Die chemische Synthese ermöglicht den Einbau von DNA-Nukleotiden als Überhänge, wodurch der siRNA-Duplex stabiler gegen Nuklease-Abbau wird.
Features and Benefits
- Alle siRNAs werden vor dem Versand MALDI-TOF MS-kontrolliert
- Renommierter siRNA-Lieferant für Top-Forschungsinstitute, Pharma-/Biotech-Unternehmen
- Ready to use: siRNA wird als gebrauchsfertige wässrige Lösung hergestellt (40 µM siRNA-Duplex, 20 mM NaCl, 10 mM TrisHCl, pH 7,5)
- Modifizierte siRNAs auf Anfrage (z. B. 2' F, 3' GalNAc)
- Einfach zu bedienendes siRNA-Design-Tool in unserem Webshop
- Sehr wettbewerbsfähige Preise
- Hohe garantierte Ausbeuten
- Ausgebildete und erfahrene Wissenschaftler unterstützen Sie gerne
- Einfach zu bedienendes Online-Portal, eine benutzerfreundliche Eingabemaske und weitere hilfreiche Tools (z. B. Auftragsverfolgung und -historie, komfortable Such- und Nachbestellmöglichkeit)
Übersicht
siRNA, oder Small Interfering Ribonucleic Acid, ist eine Art von RNA, die an einer Reihe von biologischen Prozessen beteiligt ist, vor allem an der RNA-Interferenz. RNA-Interferenz ist ein regulatorischer Prozess, der zur Kontrolle und Begrenzung der Expression bestimmter Gene eingesetzt wird. Während die meiste RNA einzelsträngig ist, besteht siRNA aus zwei komplementären Strängen von RNA-Nukleotiden, ähnlich denen in der DNA. Normalerweise sind siRNA-Moleküle 21-25 Nukleotide lange doppelsträngige RNA (dsRNA) Duplexe mit symmetrischen 2 Nukleotid 3'-Überhängen.
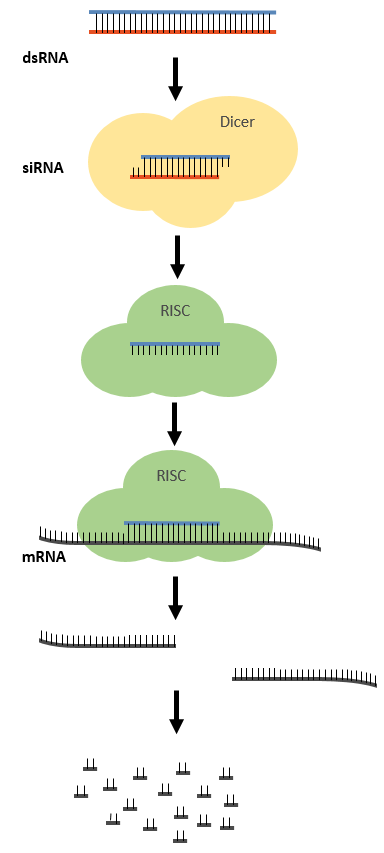
Der Prozess der RNA-Interferenz und die beteiligte biochemische Maschinerie. Doppelstrangige RNA wird durch die Endonuklease Dicer in kurze Stücke (siRNA) geschnitten. Der Antisense-Strang wird in den RISC-Komplex geladen und verbindet den Komplex durch Basenpaarung mit dem mRNA-Strang. Der RISC-Komplex schneidet den mRNA-Strang, und die mRNA wird anschließend abgebaut.
siRNA Duplexes
Microsynth liefert siRNA-Duplexe von Standard (2x21-mers) bis zu erweiterter Länge (2x27-mers) in verschiedenen Reinheitsgraden und Maßstäben, abhängig von der beabsichtigten Anwendung:
- Entsalzte siRNA (für Screening-Zwecke)
- HPLC- und PAGE-gereinigte siRNA (für Screening und für den Einsatz in Zellen)
- In vivo Tierstudien (auf Anfrage)
Produktmerkmale:
|
Synthesis scale1
|
Desalted
|
HPLC
|
PAGE
|
|||
|
OD2602
|
[nmol]2
|
OD2602
|
[nmol]2
|
[OD260] 2
|
[nmol]2
|
|
|
Genomics
|
on request
|
7
|
not available
|
not available | ||
|
0.04 µmol
|
9
|
21
|
4
|
8
|
not available
|
|
|
0.2 µmol
|
16
|
35
|
7
|
16
|
2
|
5
|
|
1.0 µmol
|
36
|
84
|
16
|
45
|
5
|
11
|
|
Higher Scales
|
on request
|
|||||
|
Classical siRNA duplexes (2x21-mers) have unit prices; if you need longer duplexes, each additional RNA base will be priced accordingly. Modified siRNAs are available upon request.
|
||||||
2 Die angegebenen Ausbeuten sind garantierte Ausbeuten und gelten für ein 2x21mer; Berechnung: 1 OD = ~2,5 nmol (bitte beachten Sie, dass diese Berechnung auf Sequenzen mit nahezu homogener Verteilung der 4 RNA-Basen basiert)
Kontroll siRNAs
Positiv- und Negativ-Kontroll-siRNAs sind eine wesentliche Ergänzung für jedes Gen-Silencing-Experiment. Positivkontrollen sind validierte siRNAs, von denen bekannt ist, dass sie hohe Knockdown-Werte (>70%) erreichen. Eine Positivkontrolle sollte verwendet werden, um die Transfektion zu optimieren und um die hohe Ausbringungsrate in jedem RNAi-Experiment zu bestätigen.
Nicht-Ziel-universelle Negativkontrollen sind validierte siRNAs, die keine Homologie zu irgendeinem bekannten Säugetiergen aufweisen. Sie sind wichtig, um sequenzspezifisches Silencing von unspezifischen Effekten im RNAi-Experiment zu unterscheiden.
Eine weitere Negativkontrollstrategie ist die Verwendung von scrambled siRNA, die die gleiche Nukleotidzusammensetzung, aber nicht die gleiche Sequenz wie die Test-siRNA aufweist. Allerdings ist es oft unmöglich, eine verschlüsselte Kontrolle zu entwerfen, die kein bekanntes Ziel in den verwendeten Zellen hat.
Im Folgenden finden Sie eine Auswahl von siRNA-Kontrollen, die in zahlreichen wissenschaftlichen Publikationen validiert wurden.
| Name | Sense Strand Sequence (5’ -3’) |
Sense Strand 3’ Overhang (5’ – 3’) |
Anti-Sense Strand 3’ Overhang (5’ – 3’) |
| Lamin | CUGGACUUCCAGAAGAACA | dTdT | dTdT |
| GFP | GCAGCACGACUUCUUCAAG | dTdT | dTdT |
| Luciferase | CGUACGCGGAAUACUUCGA | dTdT | dTdT |
| Non-targeting negative control | AGGUAGUGUAAUCGCCUUG | dTdT | dTdT |
| Scrambled negative control | On request | ||
Wie jede spezifische Ziel-siRNA können auch Kontroll-siRNAs in verschiedenen Synthesemaßstäben und Aufreinigungsstufen über den Webshop von Microsynth bestellt werden.
siRNA Design
Um Ihnen zu helfen, die am besten geeignete siRNA zu entwerfen, die auf Ihr gewünschtes Gen abzielt, hat Microsynth eine proprietäre Software entwickelt und in seinem Webshop zur Verfügung gestellt.
Das siRNA-Design-Tool von Microsynth nutzt die Richtlinien, die ursprünglich von Boese et al [1] bereitgestellt wurden. Die Ausgabe eines Designs sind verschiedene siRNAs, die nach den Reynolds-Scores [2] geordnet sind.
Die siRNA-Sequenzen mit den höchsten Scores können ausgewählt werden und direkt online bestellt werden.
Ein Link zu NCBI BLAST ist integriert, so dass Sie das Risiko möglicher Off-Target-Effekte der vorhergesagten siRNA richtig einschätzen können.
References:
[1] Boese Q, Leake D, Reynolds A, et al. Mechanistic insights aid computational short interfering RNA design. Methods Enzymol. 2005;392:73-96. doi:10.1016/S0076-6879(04)92005-8
[2] Reynolds, A., Leake, D., Boese, Q. et al. Rational siRNA design for RNA interference. Nat Biotechnol 22, 326–330 (2004). https://doi.org/10.1038/nbt936
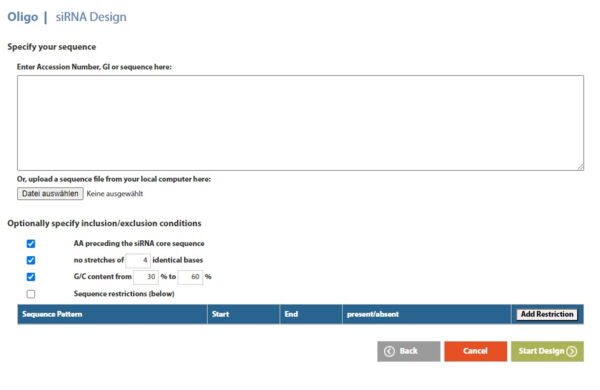
Figure: Microsynths siRNA-Design-Werkzeug
Geben Sie Ihre Zielsequenz und optional einige weitere Parameter an, wie z.B. AA vor der siRNA-Kernsequenz, die Anzahl der Abschnitte mit identischen Basen, den G/C-Gehalt sowie weitere Sequenzeinschränkungen (z.B. Sequenzbereiche, die berücksichtigt werden sollen oder nicht), um Ihren Design-Vorschlag (höchste Punktzahl oben) in einem fertigen Format zu erhalten.
Wie bestellen
- Besuchen Sie unseren Webshop
- Klicken Sie im blauen Bereich "DNA/RNA-Synthese" auf siRNA
- Wählen Sie "Normal Entry", um die gewünschten Sequenzinformationen etc. einzutippen oder zu kopieren/einzufügen, oder wählen Sie alternativ "Upload Entry", indem Sie unsere praktische Excel-Vorlage verwenden (kann bei der Bestellung heruntergeladen werden)

