Back to top
De Novo Sequenzierung

- ein Genom einer genetisch nicht charakterisierten Spezies zusammenzustellen
- unentdeckte Gene aufzuspüren
Übersicht
Überlegungen vor dem Start eines de novo Sequenzierungsprojekts:
- DNA-Qualität und vermeintliche Kontaminationen?
- Illumina oder PacBio?
- Leselänge?
- Librarygröße?
- Sequenziertiefe?
- Taxonomische Verwandtschaftsbeziehung?
- Folgestudien?
Lassen Sie sich von uns beraten - vom Design bis zur Analyse
Beispielprojekte mit de novo Sequenzierung:
- de novo Sequenzierung und Assemblierung von bisher nicht charakterisierten halophilen Bakterien
- de novo Assemblierung eines bekannten, aber stark mutierten Pilzes
- Erstellung eines Referenzgenom-Entwurfs für eine landwirtschaftlich wichtige Pflanze
- Assemblierung von ungemappten Reads innerhalb eines Resequenzierungsprojekts (neue Plasmide, große Insertionen)
- Häufige bakterielle de novo Sequenzierung
Anwendungen im Zusammenhang mit der de novo Sequenzierung:
- Reference transcriptome generation
- Resequencing
Workflow

Weitere Informationen sowie eine detaillierte technische Beschreibung finden Sie in unserer Application Note Illumina de novo Sequencing (siehe Downloads auf der rechten Seite).
Resultate
Das Hauptziel eines de-novo Assembly ist es, einzelne Reads aus NGS- oder Sanger-Sequenzierungen zu größeren Contigs und ganzen Genomsequenzen zusammenzufügen. Das Assembly ermöglicht Einblicke in die genomische Struktur eines Organismus und dessen Organisation.
Unser Assemblierungsmodul kann Reads verarbeiten, die von allen Arten von Organismen stammen, einschließlich bakterieller Genome und komplexerer eukaryotischer Organismen. Es sind sowohl de novo Ansätze als auch referenzgeführte Assemblys möglich.
Das Modul liefert die assemblierten Contigs zusammen mit Statistiken, die folgende Fragen beantworten:
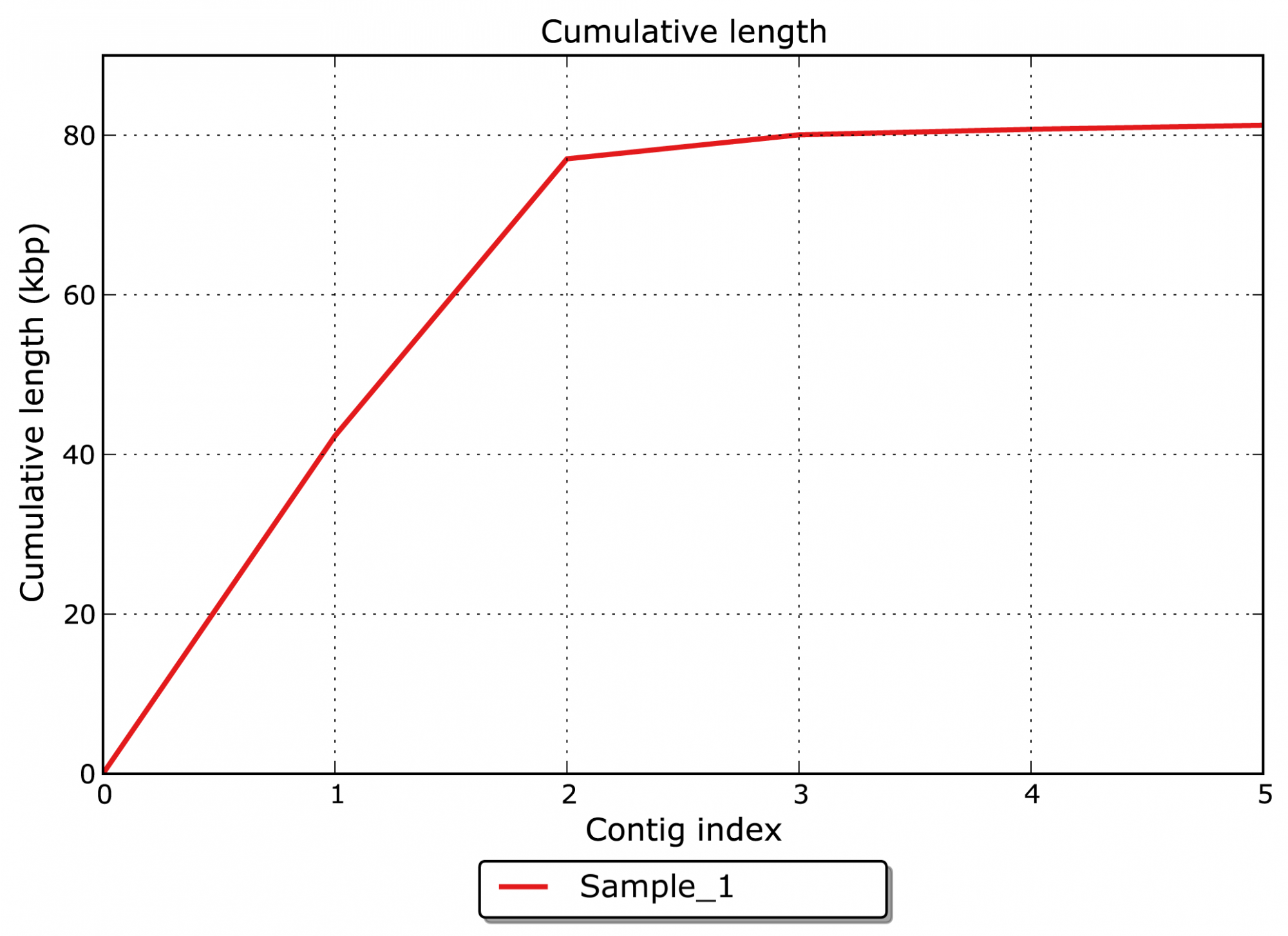
- Was ist die kumulative Gesamtlänge der assemblierten Contigs? (siehe Abbildung 1)
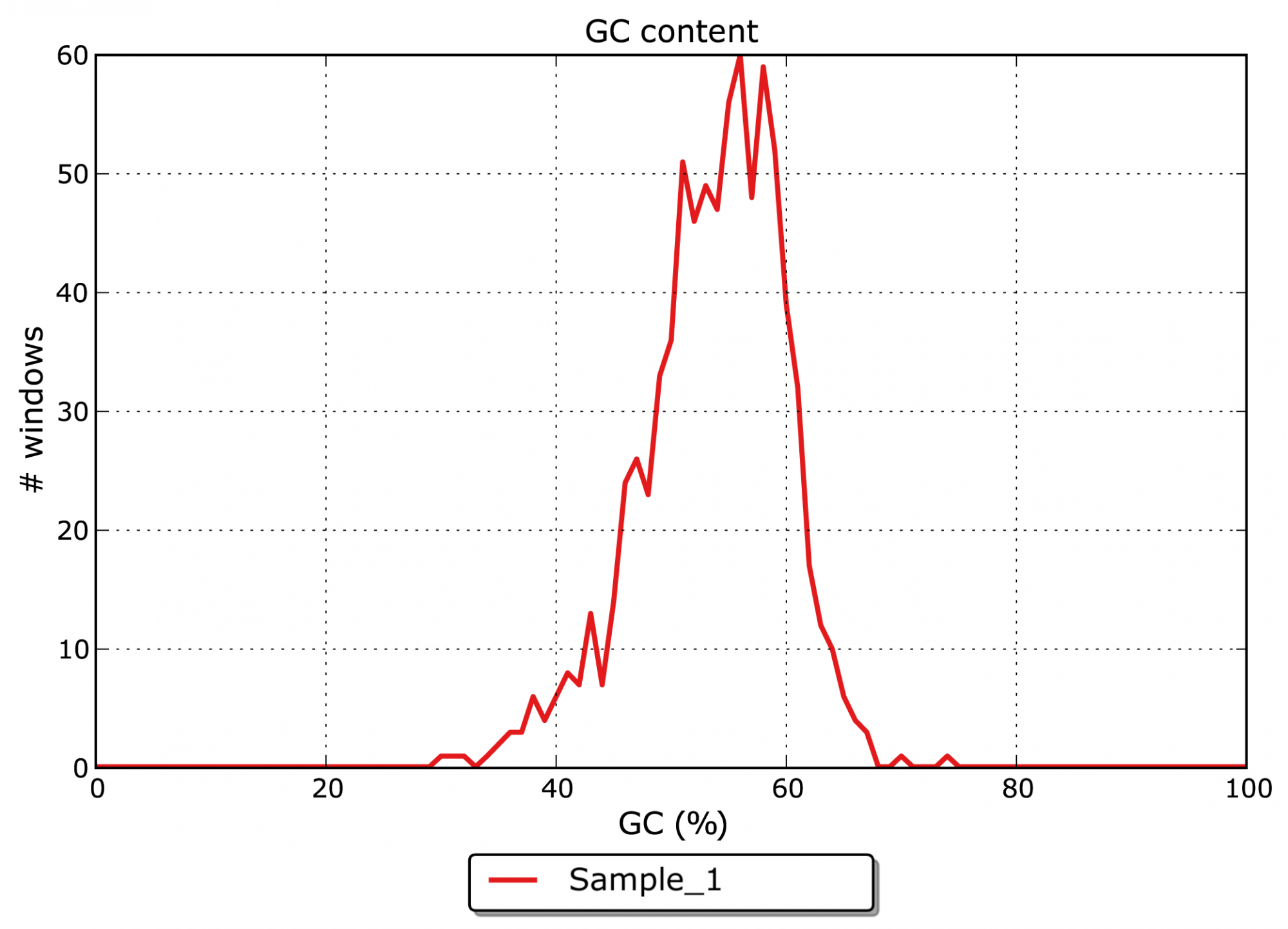
- Wie hoch ist der GC-Gehalt der assemblierten Contigs? (siehe Abbildung 2)
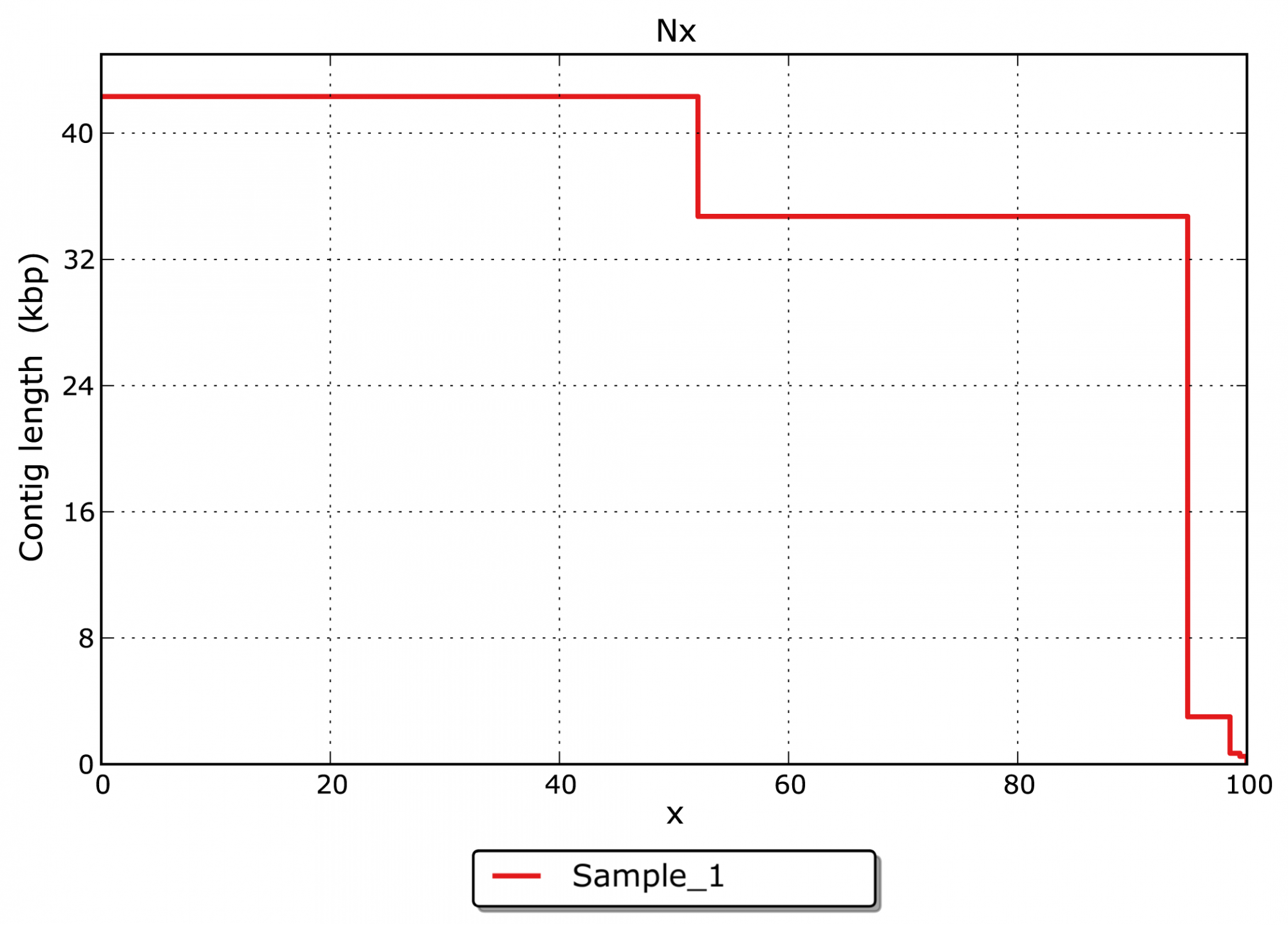
- Wie ist die Größenverteilung für die assemblierten Contigs? (siehe Abbildung 3)
Bearbeitungszeiten
- Lieferung der Daten innerhalb von 20 Arbeitstagen nach Probeneingang (inklusive Library Ertellung und Sequenzierung)
- Weitere 15 Arbeitstage für die Datenanalyse (Bioinformatik)
- Express-Service auf Anfrage möglich