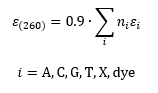Back to top
Frequently Asked Questions - Oligonukleotid Synthese
Product & Services
Ja, wir bieten MGB-Sonden mit verschiedenen Farbstoffen an.
Die Modifikation (3' MGB-Q530) kann direkt über unseren Webshop bestellt werden.
Ja, Microsynth bietet Oligos mit degenerierten Basen als Standardprodukt an. Degenerierte Basen können im Webshop direkt mit dem entsprechenden IUB-Code eingegeben werden (siehe auch Webshop). Alle degenerierten Basen (Mischbasen) werden mit hochpräzisen, vorgemischten Synthesereagenzien synthetisiert, die eine bestmögliche equimolare Verteilung der degenerierten Basen im Oligo gewährleisten.
Nein, wir empfehlen die Verwendung von Primer-BLAST für das Design Ihrer Primer.
Ja.
Microsynth Primer können in EvaGreen oder sondenbasierten Assays (z. B. Drop-off-Assays für Mutationsscreening) weiterverwendet werden.
Die bei Microsynth gekauften Thiol-modifizierten Oligonukleotide sind bereits in ihre aktive Form reduziert. Sie können die Thiol-modifizierten Oligonukleotide im gewünschten Lösungsmittel auflösen und direkt verwenden. Nach dem Auflösen empfehlen wir dringend, die Proben bei -20°C oder niedriger zu lagern.
Im Laufe der Zeit kann es nach Kontakt mit Luft oder einem Lösungsmittel, das als Oxidationsmittel wirken kann, wie z.B. DMSO, zur reversiblen Bildung einer intramolekularen Disulfidbindung kommen. In diesem Fall müssen die Thiol-modifizierten Oligonukleotide mit einem Reduktionsmittel wie DTT oder TCEP behandelt werden.
Beide Kombinationen sind möglich. Basierend auf unseren Erfahrungen zeigt HEX in Kombination mit BHQ-2 jedoch eine bessere Leistung als mit BHQ-1. Daher empfehlen wir die Verwendung von HEX in Kombination mit BHQ-2 für optimale Ergebnisse.
Fragen zur Bestellung
Um Kontroll-siRNAs zu bestellen, besuchen Sie unsere Website hier. Die aufgelisteten Sequenzen können in verschiedenen Synthesemaßstäben und Aufreinigungsgraden über den Microsynth Webshop bestellt werden.
Ja, wir können Oligos in 384-Well-Platten liefern. Bitte laden Sie unser Excel-Bestellformular unter Upload-Eintrag im Webshop herunter. Fügen Sie Ihre Sequenzen in die Datei ein und senden Sie die Excel-Datei per E-Mail an oligo.support@microsynth.ch. Schicken Sie das Excelblatt nicht direkt im Webshop ab. Wir melden uns dann bei Ihnen mit einem Angebot und weiteren Details zu Ihrer Bestellung.
Ersetzen Sie die verschiedenen DNA/RNA oder 2'-O-Methyl-RNA Basen in Ihrer Sequenz durch die internen Modifikationen 5, 6, 7 und 8 (z.B. TTAGCrAArGTTrUrC -> TTAGC5A6TT78). Loggen Sie sich in unseren Webshop ein und geben Sie Ihre Sequenz in das Sequenzfeld ein. Definieren Sie die Modifikation im Dropdown-Menü für 5, 6, 7, 8 (entweder RNA/DNA oder 2'-O-Methyl-RNA Basen).
Sie können die Position der Oligonukleotide in der 96-Well-Platte verschieben, indem Sie eine Leerposition setzen. Geben Sie dazu einen Oligo-Datensatz mit den folgenden Eigenschaften ein:
i. Oligoname: Leerposition
ii. Scale: Genomics
iii. Purification: Desalted
iv. Sequence: TT
Die Mindestmenge an Oligos, um eine 96-Well-Platte zu erhalten, beträgt 40 Oligonukleotide.
- Wählen Sie ein "T" DNA-Nukleotid innerhalb Ihrer Sequenz und ersetzen Sie das "T" durch "5".
- Wählen Sie Ihren bevorzugten Farbstoff (z. B. FAM-dT) im Dropdown-Menü unter "Innere Modifikation (5=...)" aus
- Folgen Sie den weiteren Anweisungen und senden Sie Ihre Bestellung ab
Erstes Screening mit verschiedenen siRNAs: Für eine solche Anwendung verwenden unsere Kunden in der Regel entsalzte siRNA, die einer Kartuschenreinigung unterzogen wurde. Damit werden Reinheitsgrade von ~80 % erreicht, was für erste Screening-Zwecke völlig ausreichend ist.
Fortgeschrittenes Screening oder für den Einsatz in Zellkulturen: wenn Ihr Experiment anspruchsvoller und damit teurer wird, empfehlen wir in der Regel die HPLC- (~85 %) oder sogar PAGE-Aufreinigung (~95 %).
Für größere Mengen, ab 5 mg, bieten wir siRNAs mit niedrigem Endotoxingehalt an, die speziell für den Einsatz in Tierversuchen geeignet sind. Bitte kontaktieren Sie uns für weitere Informationen.
Standardmäßig befindet sich an der 5' und 3' Seite eines Oligos eine OH-Gruppe.
Molare Extinktionskoeffizienten von Oligonukleotiden werden nach dem "Base Composition Model" berechnet. [1-3] Es beinhaltet die Absorption der einzelnen Nukleobasen und mögliche Basenmodifikationen und/oder Farbstoffe. Genauere Werte für unmodifizierte Oligonukleotide erhält man mit dem "nearest-neighbor model", das Stapelungseffekte von benachbarten Nukleobasen berücksichtigt. Nach unseren Berechnungen reduziert dieses Modell den Extinktionskoeffizienten um ca. 90 % im Vergleich zum Basenzusammensetzungsmodell. Daher werden die Extinktionskoeffizienten von Oligonukleotiden nach der folgenden Gleichung geschätzt.
- Tataurov A.V., You Y., and Owczarzy R. (2008) Predicting ultraviolet spectrum of single stranded and double stranded deoxyribonucleic acids, Biophys. Chem. 133, 66-70.
- Fasman, G.D. (Ed.) (1975) Handbook of Biochemistry and Molecular Biology, Volume 1: Nucleic Acids, pp 589, 3rd edition, CRC Press.
- Cavaluzzi M.J., and Borer P.N. (2004) Revised UV extinction coefficients for nucleoside-5'-monophosphates and unpaired DNA and RNA, Nucleic Acids Res. 32, e13.