Back to top
Shotgun Metatranscriptomics

- die aktive genetische Zusammensetzung Ihrer Proben zu untersuchen.
- einen funktionellen Einblick in die bakterielle Gemeinschaft zu erhalten.
- aktive Transkripte und hoch- oder herunterregulierte Stoffwechselwege zu entdecken.
Übersicht
Überlegungen vor dem Start eines Shotgun-Metatranskriptomik-Projekts:
- Menge und Herkunft der Proben?
- Angemessene Probenahmetechnik?
- Sequenzierungstiefe (Empfindlichkeit)?
- Deskriptive und/oder empirische Studie?
- Fokus auf Genexpression, Taxonomie, funktionelles Potenzial?
- Komplexität und Zusammensetzung der Gemeinschaft?
Lassen Sie sich von uns beraten - vom Entwurf bis zur Analyse
Beispielprojekte, die Shotgun-Metatranskriptomik verwenden:
- Aktivierte Enzymkaskaden unter Bioreaktorbedingungen
- Funktionelle regulatorische Interaktionen zwischen Wirt und Pathogen
- Entdeckung neuer enzymatischer Interaktionen
- Evolutionäre Anpassungen in Mikroumgebungen
- Genexpressionsanalyse nicht kultivierbarer bakterieller Gemeinschaften
Anwendungen im Zusammenhang mit Shotgun Metatranskriptomik:
- RNA sequencing
- Whole genome metagenomics
- Amplicon metagenomics
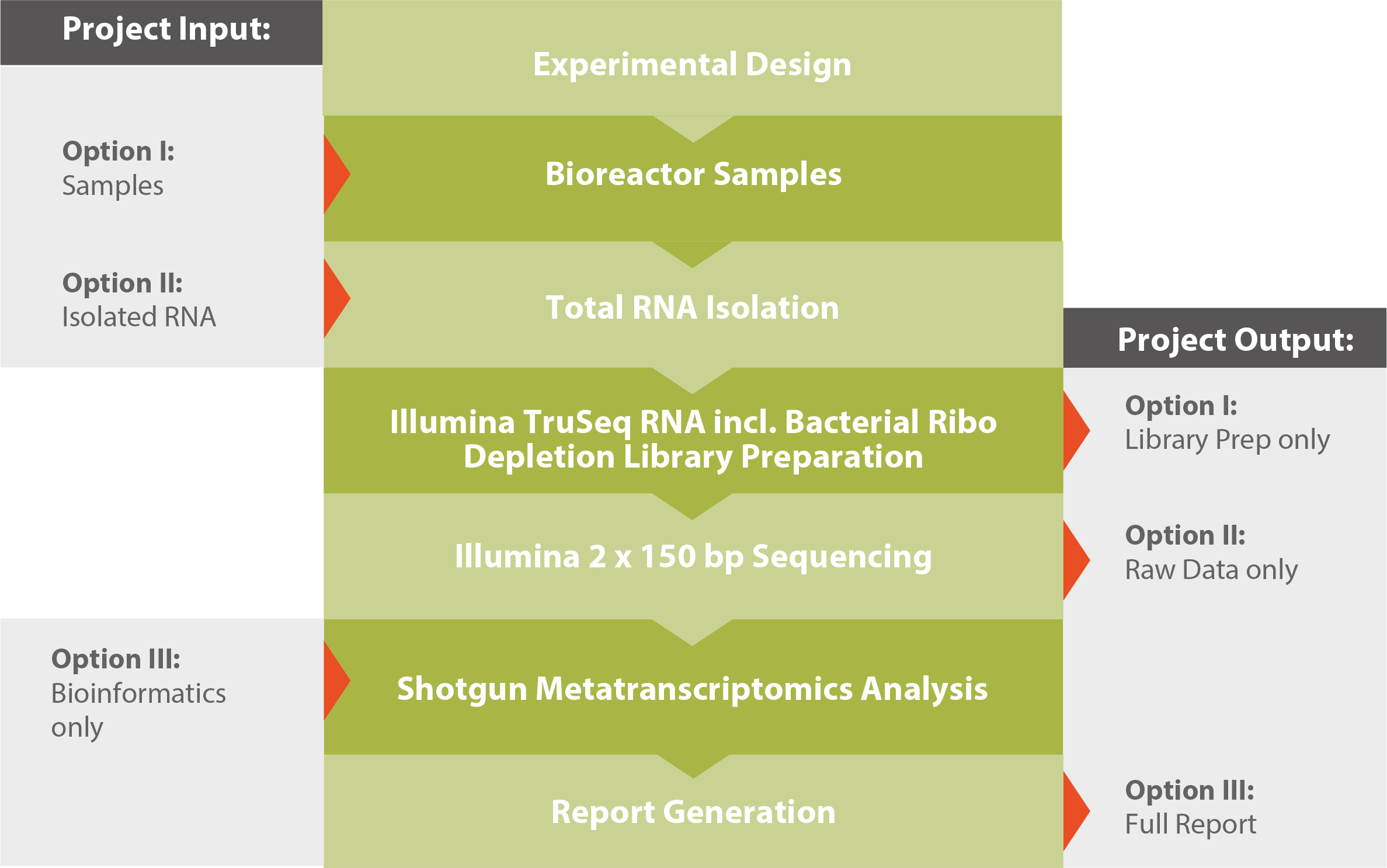
Workflow
Resultate
Die Shotgun Metatranskriptomik-Analyse basiert auf RNA, die aus einer mikrobiellen Gemeinschaft extrahiert wird. Während sich die Shotgun-Metagenomik auf die taxonomische Zusammensetzung und das funktionelle Potenzial einer Gemeinschaft konzentriert, befasst sich die Shotgun-Metatranskriptomik mit dem Teil der Gemeinschaft, der aktiv Gene exprimiert, und beantwortet diese Hauptfragen:
- Welche Gene in der mikrobiellen Gemeinschaft werden aktiv exprimiert? (siehe Tabelle 1)
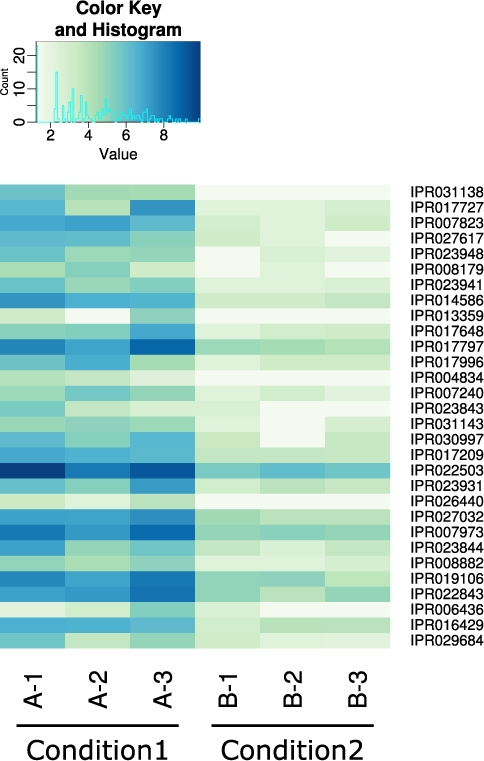
- Welche taxonomischen Gruppen sind Teil der aktiven mikrobiellen Gemeinschaft? (siehe Abbildung 1)
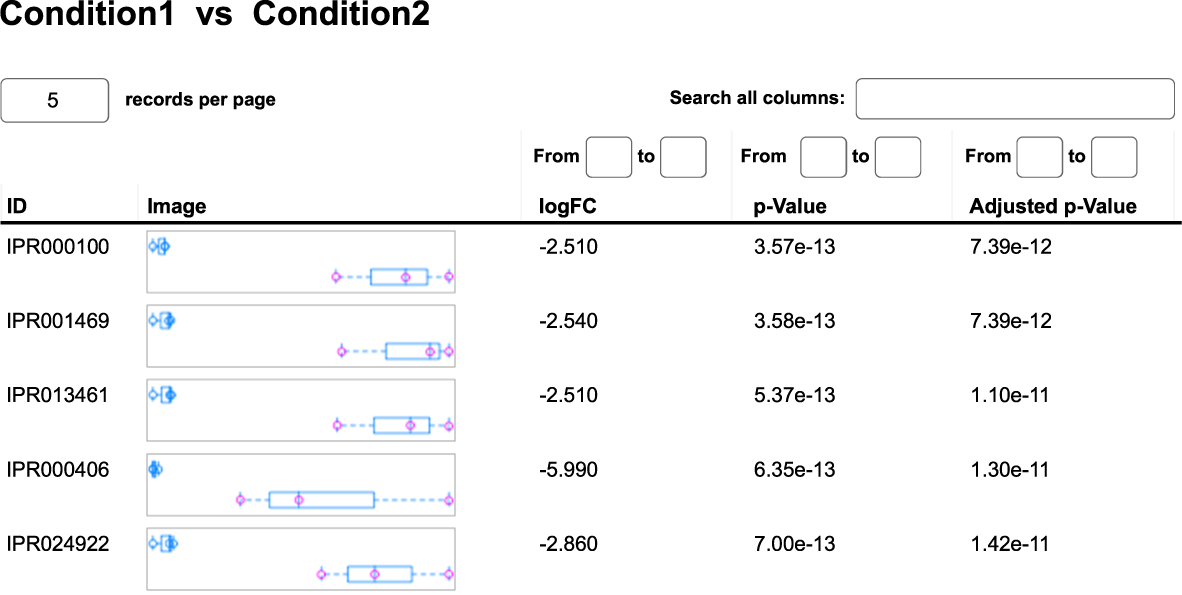
- Gibt es Unterschiede in der Expression von funktionellen Genen zwischen Gemeinschaften oder Bedingungen? (siehe Abbildung 2)
Mit unserem Analysemodul erhalten Sie einen vollständigen Überblick über die exprimierten funktionellen Gene und die taxonomische Zusammensetzung der aktiven mikrobiellen Gemeinschaft. Zusätzlich können unterschiedlich exprimierte Gene und Merkmale zwischen verschiedenen Bedingungen oder unterschiedlichen Gemeinschaften identifiziert werden.
Tabelle 1: Zusammenfassende Tabelle, die sich aus der differenziellen Genexpressionsanalyse des metatranskriptomischen Datensatzes ergibt. Die IDs beziehen sich auf die InterPro-Klassifikation für Proteine. Die Spalte logFC steht für die logarithmische Fold Change zwischen Condition1 und Condition2. Die statistische Signifikanz wird durch den p-Wert und den bereinigten p-Wert für Mehrfachtests angegeben.
Bearbeitungszeiten
- Delivery of data within 30 working days upon sample receipt (includes library preparation and sequencing)
- Additional 10 working days for data analysis (bioinformatics)
- Express service possible on request