Back to top
Métatranscriptomique Shotgun

- Étudier la composition génétique active de vos échantillons
- Obtenir un aperçu fonctionnel au niveau de la communauté bactérienne
- Découvrir les transcriptions actives et les voies de régulation amplifiées ou diminuées
Aperçu
Considérations avant de lancer un projet de métatranscriptomique shotgun :
- Quantités d'échantillons et origine ?
- Technique d'échantillonnage adéquate ?
- Profondeur de séquençage (sensibilité) ?
- Étude descriptive et/ou empirique ?
- Accent mis sur l'expression des gènes, la taxonomie, le potentiel fonctionnel ?
- Complexité et constitution de la communauté ?
Laissez-nous vous guider - de la conception à l'analyse
Exemples de projets utilisant la métatranscriptomique shotgun :
- Cascades enzymatiques activées dans des conditions de bioréacteur
- Interactions de régulation fonctionnelles entre l'hôte et l'agent pathogène
- Découverte de nouvelles interactions enzymatiques
- Adaptations évolutives dans les micro-environnements
- Analyse de l'expression génétique de communautés bactériennes non cultivables
Applications liées à la métatranscriptomique shotgun :
- Séquençage de l'ARN
- Métagénomique shotgun
- Métagénomique des amplicons
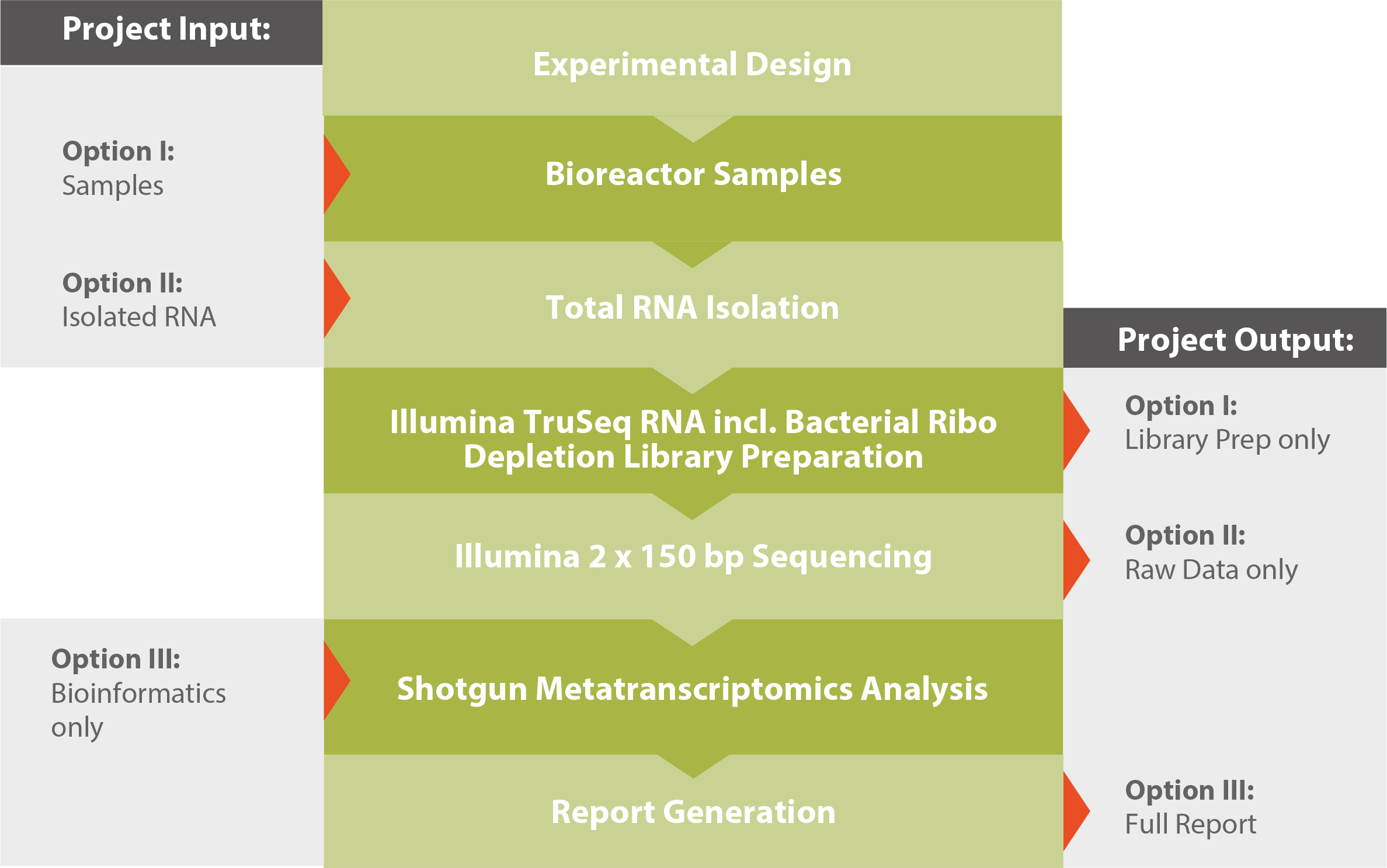
Déroulement
Résultats
L'analyse métatranscriptomique shotgun est basée sur l'ARN extrait d'une communauté microbienne. Alors que la métagénomique shotgun se concentre sur la composition taxonomique et le potentiel fonctionnel d'une communauté, la métatranscriptomique shotgun s'intéresse à la partie de la communauté qui exprime activement les gènes et répond à ces principales questions :
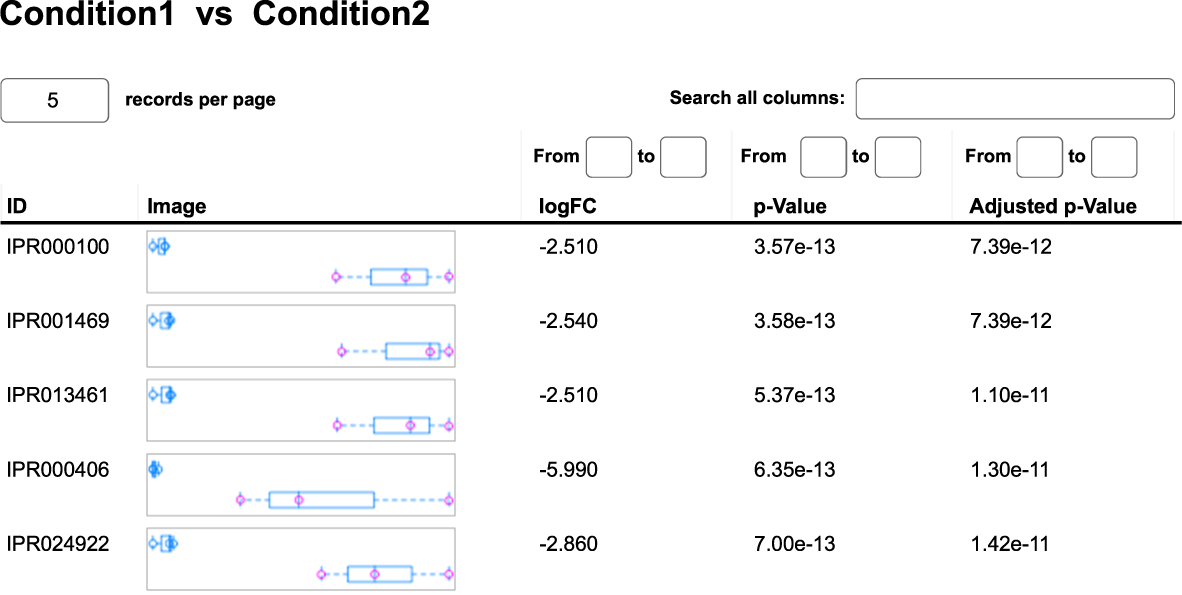
- Quels gènes de la communauté microbienne sont activement exprimés ? (voir tableau 1)
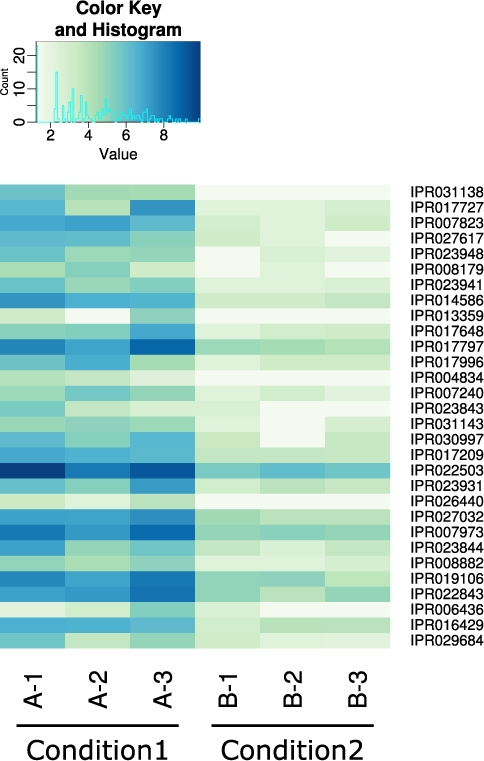
- Quels sont les groupes taxonomiques qui font partie de la communauté microbienne active ? (voir figure 1)
- Y a-t-il des différences dans l'expression des gènes fonctionnels entre les communautés ou les conditions ? (voir figure 2)
Notre module d'analyse permet d'obtenir une vue d'ensemble complète des gènes fonctionnels exprimés et de la composition taxonomique de la communauté microbienne active. En outre, les gènes et les caractéristiques exprimés de manière différentielle peuvent être identifiés entre des conditions variables ou des communautés différentes.
Table 1: Tableau récapitulatif résultant de l'analyse de l'expression génétique différentielle de l'ensemble des données métatranscriptomiques. Les ID font référence à la classification InterPro pour les protéines. La colonne logFC représente le changement de fold logarithmique entre la condition 1 et la condition 2. La significativité statistique est donnée par la p value et la valeur p ajustée pour les tests multiples.
Délai d'exécution
- Livraison des données dans les 30 jours ouvrables suivant la réception de l'échantillon (comprend la préparation et le séquençage de la bibliothèque)
- 10 jours ouvrables supplémentaires pour l'analyse des données (bioinformatique)
- Service express possible sur demande