Back to top
Séquençage de l'Ensemble de l'Exome

Utilisez le séquençage de l'exome entier pour :
- Se concentrer sur des régions sélectionnées et de petites variations au sein d'un génome
- Limiter les observations aux régions codant pour les protéines d'un génome dans un contexte de diagnostic
- Réduire les coûts et la durée pour la détection récurrente dans une étude à long terme
Aperçu
Considérations avant de commencer un projet de séquençage de l'exome entier :
- Quel organisme et quel génome de référence ?
- Objectif de l'étude ?
- Couverture requise ?
Laissez-nous vous guider - de la conception à l'analyse
Exemples de projets utilisant le séquençage des exomes entiers :
- Observations des variations de nucléotides simples (SNV) et des petites insertions et délétions (InDels) pour les échantillons de cancer
- Comparaison des profils des SNV
- Observation au cours du temps des conséquences du traitement mutagène sur la modification des protéines
Applications liées au séquençage des exomes entiers :
- Reséquençage eucaryote
- Développement de marqueurs microsatellites
Déroulement
Le graphique ci-dessous illustre un déroulement typique d'un projet de séquençage d'exome. Veuillez noter que nos processus organisés en modules vous offrent diverses options d'entrée et de sortie. Vous pouvez externaliser la totalité ou une partie de votre projet NGS à Microsynth.
Résultats
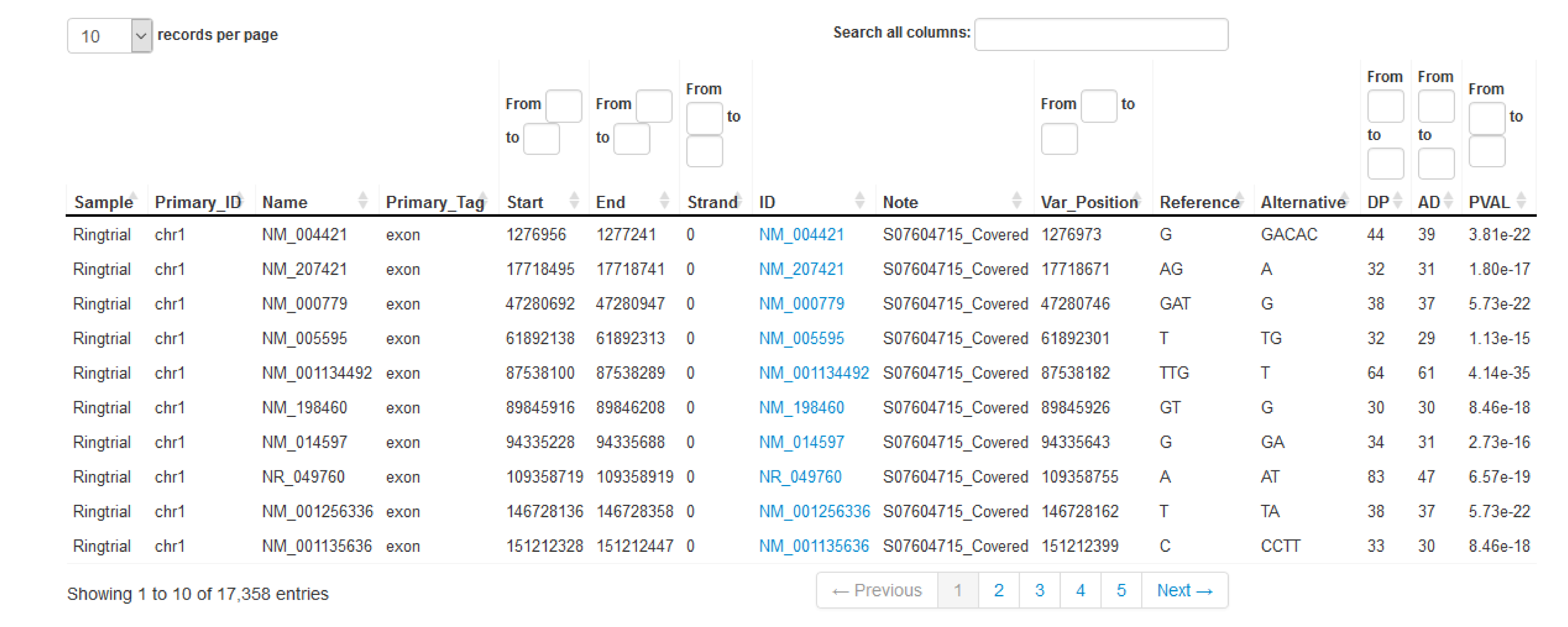
Les résultats produits par notre module d'analyse permettent de répondre à deux questions principales d'un reséquençage d'un exome eucaryote visant à détecter des variations par rapport à un exome de référence pré-annoté.
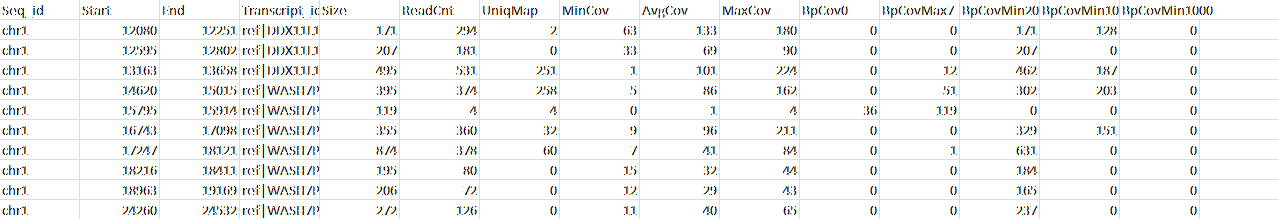
- Quelle était la couverture de l'exome cible ? (voir tableau 1)
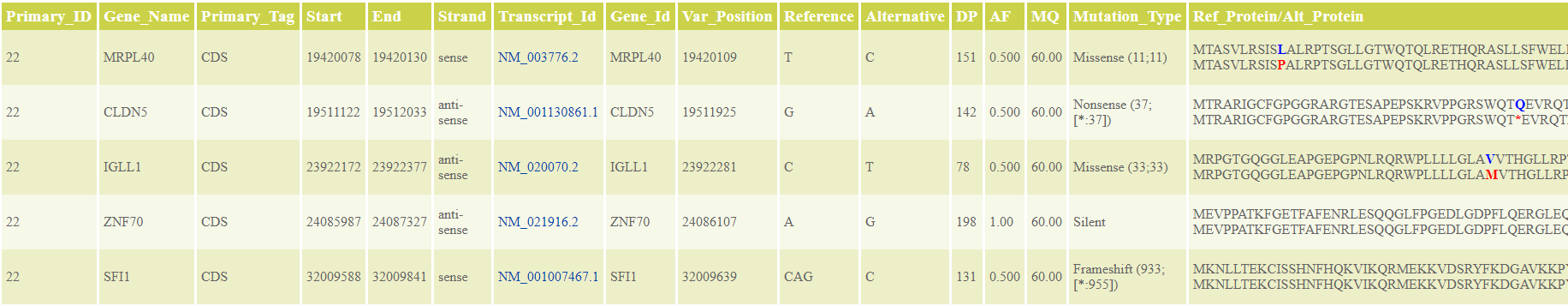
- Quelles sont les variations des nucléotides simples et les petites insertions/délétions par rapport au génome de référence et quels sont les effets de ces variations détectées au niveau des protéines ? (voir tableaux 2A et 2B)
- Parmi les variations détectées, lesquelles sont également connues et répertoriées dans les bases de données publiques (par exemple, ClinVar - NCBI - NIH) ? (voir tableau 3)
Table 1: Ceci est un extrait du tableau complet détaillant la couverture de reads pour chaque cible dans l'exome séquencé.
Délai d'exécution
- Livraison des données dans les x jours ouvrables suivant la réception de l'échantillon (comprend la préparation et le séquençage de la bibliothèque)
- x jours ouvrables supplémentaires pour l'analyse des données (bioinformatique)
- Service express possible sur demande
x= sur demande