Back to top
Sondes qPCR et dPCR sur Mesure

Augmentez la sensibilité et la spécificité de votre PCR en temps réel ou digitale avec les sondes de Microsynth, de haute qualité avec une large gamme de dyes et de quenchers sont livrées à une vitesse exceptionnelle.
Caractéristiques et Avantages
- Procédés de purification poussés (HPLC ou même PAGE)
- Un contrôle qualité rigoureux (surveillance online du trityl et MALDI-TOF MS)
- Délais de 3 à 5 jours ouvrables
Système Simple de Commande en Ligne
-
Site en ligne facile à utiliser, doté d'une série d'outils utiles (par exemple, suivi et historique des commandes, option de recherche et de commande pratique)
- Possibilité d'ajuster l'affinité de liaison via MGB, LNA et autres amplificateurs Tm, ou même de combiner différents amplificateurs Tm pour personnaliser votre dosage.
- Grand choix de combinaisons fluorophores - quenchers
- Service de conception professionnelle
- Processus de production certifié EN ISO 13485:2016
- Le développement, la validation, la production et les tests des dosages qPCR et PCR numériques peuvent être externalisés chez Microsynth.
- Des scientifiques formés et expérimentés sont heureux de vous aider.
Sondes Dual Labeled
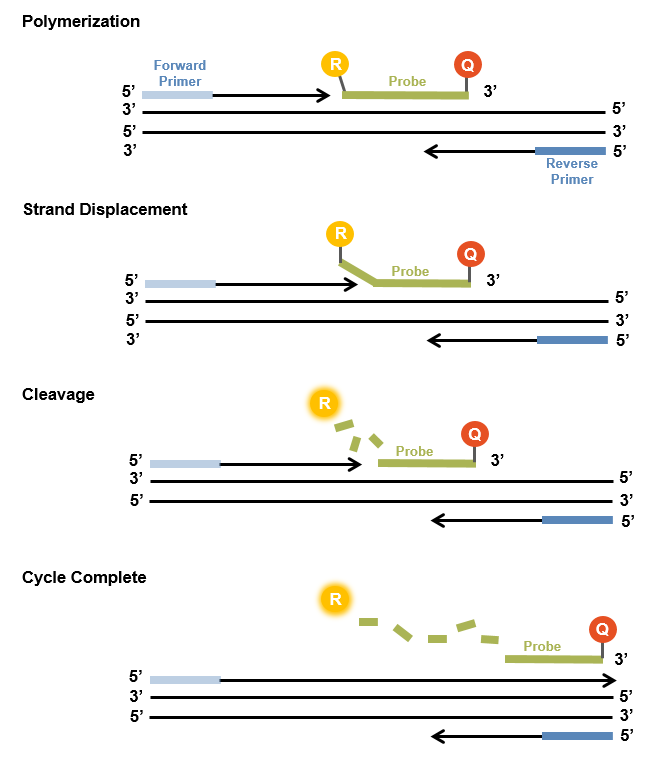
Les sondes dites TaqMan sont des sondes d'hydrolyse qui sont conçues pour augmenter la spécificité de la PCR quantitative.
À l'état hybridé, la proximité du fluorophore et du quencher est suffisante pour éteindre la fluorescence. Pendant la PCR, l'activité exonucléase 5'-3' de la Taq polymérase est utilisée pour cliver la sonde à double marquage. Le fluorophore et le quencher sont ainsi séparés et un signal de fluorescence permet une mesure quantitative.
La sonde TaqMan augmente considérablement la spécificité de la détection.
Nous serions heureux de vous aider à concevoir les sondes et les amorces associées.
| 5‘ Label | 3‘ Label | 0.04 µmol scale1,2,3 | 0.2 µmol scale1,2,3 | 1.0 µmol scale1,2,3 |
|
FAM |
BHQ-1 |
1 OD |
2 OD 10 nmol |
12 OD 60 nmol |
|
AlexaFluor Dyes |
BHQ-1 |
0.75 OD |
1.5 OD 7.5 nmol |
8 OD 40 nmol |
|
HEX |
BHQ-2 |
1 OD |
2 OD 10 nmol |
12 OD 60 nmol |
|
AlexaFluor Dyes |
BHQ-2 | 0.75 OD 3.75 nmol |
1.5 OD 7.5 nmol |
8 OD 40 nmol |
2 Les rendements indiqués dans la DO 260 s'appliquent à une sonde composée de 20 bases d'ADN ; Calcul : 1 DO = 5 nmol (veuillez noter que ce calcul est basé sur des séquences ayant une distribution pratiquement homogène des 4 bases d'ADN ; il peut varier pour des séquences ayant une teneur élevée en GC >70 %, etc.)
Double-Quenched Probes
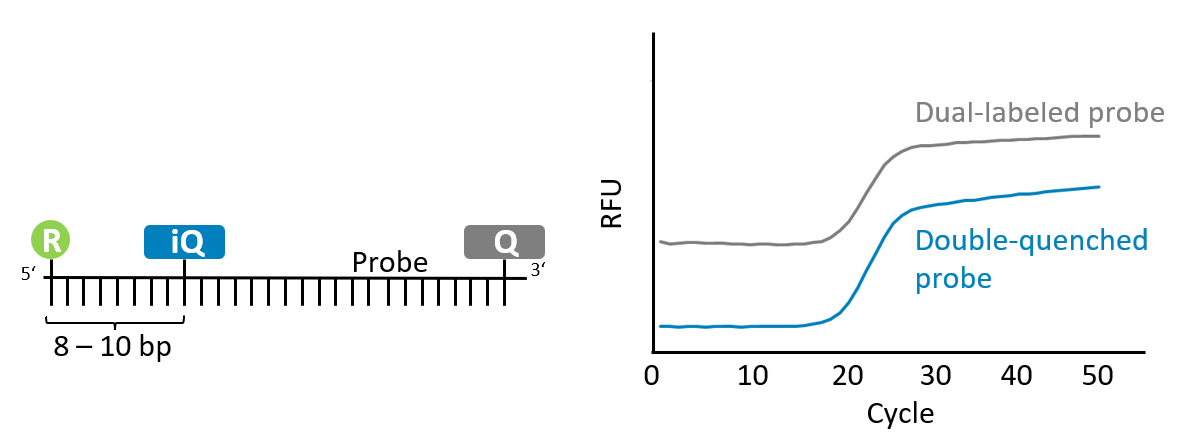
Les sondes qPCR standard pour les essais de 5'-nucléase utilisent généralement un fluorophore à l’extrémité 5' et un extincteur (quencher) à l’extrémité 3' pour supprimer la fluorescence et empêcher l’extension de la sonde pendant la PCR. Cependant, lorsque la longueur de la sonde augmente, le signal de fond tend souvent à s’intensifier en raison de la distance accrue entre le fluorophore et l’extincteur.
Les sondes à double extinction offrent une solution supérieure en réduisant la fluorescence de fond et en améliorant la sensibilité. En intégrant un extincteur supplémentaire à 8–10 bases du fluorophore 5', elles augmentent l’efficacité du transfert d’énergie et offrent un rapport signal-bruit nettement amélioré. Ce design est particulièrement avantageux pour les sondes longues, prenant en charge des longueurs allant jusqu’à 40 nucléotides.
Notre large gamme de sondes à double extinction
Microsynth propose une variété de sondes à double extinction adaptées à vos besoins spécifiques, notamment :
- Combinaisons avancées fluorophore-extincteur conçues pour des applications exigeantes, telles que les dosages multiplex ou les cibles complexes.
Nous serions ravis de vous accompagner dans la conception des sondes et des amorces associées.
| 5‘ Dye | 3‘ Quencher | Internal Quencher | 0.04 µmol scale1,2 | 0.2 µmol scale1,2 | 1.0 µmol scale1,2 |
| FAM | BHQ-1 or TAMRA | iQ530 | 1 OD 5 nmol |
2 OD 10 nmol |
8 OD 40 nmol |
| Yakima Yellow | BHQ-1 | iQ530 | 1 OD 5 nmol |
2 OD 10 nmol |
8 OD 40 nmol |
| HEX | BHQ-2 | iQ530 | 1 OD 5 nmol |
2 OD 10 nmol |
8 OD 40 nmol |
| ROX | BHQ-2 | iQ530 | 0.75 OD 3.75 nmol |
1.5 OD 7.5 nmol |
8 OD 40 nmol |
| CalRed | BHQ-2 | iQ530 | 0.75 OD 3.75 nmol |
1.5 OD 7.5 nmol |
8 OD 40 nmol |
| Cy5 | BHQ-2 | iQ530 | 0.75 OD 3.75 nmol |
1.5 OD 7.5 nmol |
8 OD 40 nmol |
| A647N | BHQ-2 | iQ530 | 0.75 OD 3.75 nmol |
1.5 OD 7.5 nmol |
8 OD 40 nmol |
1 L'échelle de synthèse représente la quantité initiale de bases 3' (matériel de départ).
2 Les rendements indiqués dans la DO 260 s'appliquent à une sonde composée de 20 bases d'ADN ; Calcul : 1 DO = 5 nmol (veuillez noter que ce calcul est basé sur des séquences ayant une distribution pratiquement homogène des 4 bases d'ADN ; il peut varier pour des séquences ayant une teneur élevée en GC >70 %, etc.)
Applications Potentielles
- Des applications de qPCR exigeantes avec la nécessité d’une plus grande flexibilité dans la conception des séquences, par exemple pour les cibles riches en AT et nécessitant des sondes plus longues (≥25 bases)
- L'analyse ddPCR où la sonde doublement marquées facilite la discrimination des gouttelettes positives par rapport aux gouttelettes négatives (cluster amélioré)
LNA Probes
Le Locked Nucleic Acid (LNA) s'est révélé être un outil puissant dans de nombreuses applications de qPCR et de PCR digitale. Lorsqu'il est incorporé dans des oligonucléotides d'ADN il offre les avantages suivants par rapport aux bases d'ADN à l'état natif uniquement :
Stabilité thermique et spécificité d'hybridation accrues
Une quantification plus précise des gènes et une discrimination allélique
Des designs plus simples et plus modulables pour les séquences cibles problématiques
Chaque ajout de base LNA dans un oligo augmente la Tm d'environ 2-4 °C et les sondes d'acide nucléique avec LNA peuvent être conçues pour avoir un ΔTm de >15°C. Habituellement, pas plus de 5 bases LNA sont incorporées dans la séquence d'une sonde. Lors de la conception et de l'optimisation de vos sondes LNA, veuillez suivre les recommandations décrites ici.
Chez Microsynth, vous pouvez commander votre sonde LNA avec la gamme complète de dyes et de quenchers que nous proposons également pour nos sondes à double marquage.
| 0.04 µmol scale1,2 | 0.2 µmol scale1,2 | 1.0 µmol scale1,2 | ||
| LNA probes |
0.5 OD |
1 OD 5 nmol |
6 OD 30 nmol |
|
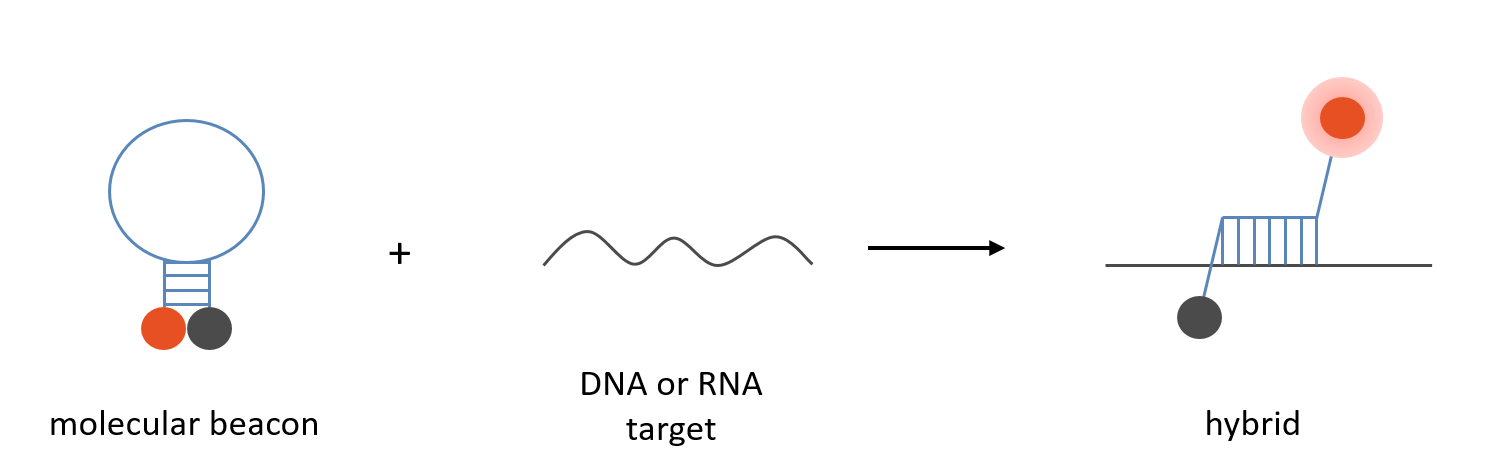
Les Molécules Beacons
Les moléculaires beacons sont des sondes d'hybridation d'oligonucléotides simple brin qui forment une structure en tige-boucle. La boucle contient la séquence de la sonde complémentaire à une séquence cible, et la tige est formée par annealing de séquences de bras complémentaires qui sont situées de part et d'autre de la séquence de la sonde. Par conséquent, un fluorophore à l'extrémité 5' et un quencher à l'extrémité 3' sont à proximité immédiate et ne sont pas fluorescents. Cependant, lorsqu'ils s'hybrident à un brin d'acide nucléique contenant une séquence cible, ils subissent un changement de conformation qui leur permet d'être fluorescents.
Les moléculaires beacons sont techniquement traitées comme des sondes à double marquage. Les mêmes spécifications et rendements s'appliquent.
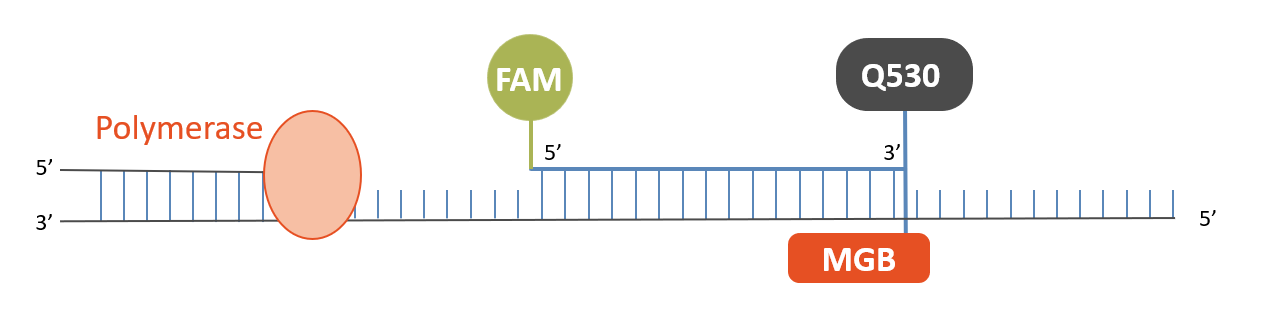
MGB Probes
Les sondes d'ADN conjuguées au minor groove binder (MGB) forment des duplex exceptionnellement stables avec des cibles d'ADN simple brin, ce qui permet d'utiliser des sondes plus courtes pour des analyses basés sur l'hybridation.
- une plus grande sélectivité de liaison à la cible
- moins de fluorescence de fond
- une meilleure qualité de la sonde grâce à une longueur plus courte
| 5‘ Label | 3‘ Label | Channel (Typical) | 0.04 µmol scale1,2,3 | 0.2 µmol scale1,2,3 | 1.0 µmol scale1,2,3 |
| FAM | MGB-Q530 | Channel 1 / Green |
1 OD
5 nmol
|
2 OD
10 nmol
|
10 OD
50 nmol
|
| JOE / HEX / Yakima Yellow |
MGB-Q530 | Channel 2 / Yellow | |||
| California Red | MGB-Q530 | Channel 3 / Red | |||
| Atto647N | MGB-Q530 | Channel 4 / Deep Red |
2 Les rendements indiqués dans la DO 260 s'appliquent à une sonde composée de 20 bases d'ADN ; Calcul : 1 DO = 5 nmol (veuillez noter que ce calcul est basé sur des séquences ayant une distribution pratiquement homogène des 4 bases d'ADN ; il peut varier pour des séquences ayant une teneur élevée en GC >70 %, etc.)
Service de Design
Designs Simples
Les tests avec sonde à double marquage sont les plus courants dans le domaine de la PCR en temps réel. Les tests avec sonde à double marquage sont généralement plus spécifiques que les tests SYBR Green. La mise en place d'un test avec sonde nécessite également moins d'étapes d'optimisation et les résultats de l'analyse sont plus faciles à interpréter. En utilisant des sondes bien conçues et de haute qualité dans votre analyse, vous réduisez le coût de l'optimisation de l'essai et les éventuelles corrections d'erreurs et répétitions de vos mesures par rapport aux essais SYBRGreen, ce qui rend votre analyse plus rapide et plus rentable.
Grâce à la disponibilité de divers outils logiciels gratuits en ligne, une bonne conception d'analyse simpleplex impliquant une sonde à double marquage (également appelée hydrolyse ou sonde TaqMan) est facile à réaliser et ne nécessite pas de connaissances détaillées (il suffit de suivre les principales recommandations).
Microsynth recommande les outils de design suivants qui facilitent la conception d'amorces et de sondes à double marquage hautement personnalisées pour les applications de routine de qPCR :
- Primer-BLAST
Primer-BLAST a été développé au NCBI pour aider les utilisateurs à la conception d'amorces spécifiques à la cible. Il utilise Primer3 pour concevoir des amorces PCR, puis utilise BLAST et un algorithme d'alignement global pour passer les amorces au crible selon la base de données sélectionnée par l'utilisateur afin d'éviter les paires d'amorces (toutes les combinaisons, y compris les paires d'amorces forward-forward, forwardt-reverse et reverse-reverse) qui peuvent provoquer des amplifications non spécifiques. Ye J, Coulouris G, Zaretskaya I, Cutcutache I, Rozen S, Madden T (2012). Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction. BMC Bioinformatics. - Primer3Plus
Steve Rozen and Helen J. Skaletsky (2000) Primer3 on the WWW for general users and for biologist programmers. In: Krawetz S, Misener S (eds) Bioinformatics Methods and Protocols: Methods in Molecular Biology. Humana Press, Totowa, NJ, pp 365-386
Design Plus Complexes (sur demande)
Lorsque vous planifiez des expériences nécessitant un design plus complexe telles l'épissage, le multiplexage, SNP et CNV, ou de chimie spéciale (LNA/MGB/Propynyl), vous pouvez compter sur l'expérience de Microsynth dans ce domaine (>2 décennies) ainsi que sur des outils de conception professionnels sélectionnés pour des applications aussi exigeantes.
Si vous avez besoin d'une aide supplémentaire pour la conception d'amorces et de sondes, contactez-nous à l'adresse myproject@microsynth.ch. N'oubliez pas de préciser les points suivants :
- Objectif expérimental (analyse de l'expression, détection de l'agent pathogène, etc.)
- Numéro(s) d'accession ou, à défaut, les séquences de gènes de votre cible
- Échelle de synthèse souhaitée ainsi que le marquage 5' et 3'
Comment Commander
- Entrez dans notre boutique en ligne
- Cliquez sur l'ADN dans le domaine " DNA/RNA synthesis"
- Sélectionnez soit Normal Entry pour écrire ou copier/coller les informations de la séquence souhaitée, etc. soit Upload Entry en utilisant notre template Excel pratique (peut être téléchargé lors de la commande)
- Sondes single-quenched (sondes TaqMan, Molecular Beacon, MGB et LNA) : Sélectionnez la modification 5', 3' souhaitée ainsi que la purification HPLC, votre oligo est reconnu et traité en tant que sonde single-quenched dans le cas des sondes LNA, incluez les symboles de commande "5, 6, 7 et 8" dans votre séquence et choisissez "LNA-A, LNA-C, LNA-G et LNA-T" sous "Modification interne (5=...)", etc.
Sondes double-quenched: insérez le symbole de commande "5" dans votre séquence (par exemple catattgaa5actgggttaacggaatt) et choisissez "Internal Quencher 500" sous Inner Modification ("5=..."). Sélectionnez la modification 5', 3' souhaitée ainsi que la purification HPLC, votre oligo est reconnu et traité comme une sonde double-quenched.
- Suivez ensuite les instructions
1 Microsynth fournit des sondes qPCR de la plus haute qualité. La majorité des sondes PCR de Microsynth sont purifiées par HPLC, tandis que quelques-unes nécessitent une purification par PAGE afin de répondre aux spécifications strictes du contrôle qualité. Si votre combinaison Dye/quencher n'est pas compatible avec l'HPLC, utilisez simplement la PAGE.