
Back to top
Analyse Epidémiologie Génomique

Utilisez le service d'analyse d'épidémiologie génomique de Microsynth pour :
- L'assemblage et annotation de-novo de génomes
- Screening de la présence de gènes de virulence, de résistance et de mycotoxine
- Typage de plusieurs souches bactériennes simultanément (par exemple, analyse cgMLST)
- Déterminer la composition des méta-communautés
- Détecter les variations génétiques et étudier leurs conséquences
Aperçu
Considérations avant de commencer un projet d'analyse d'épidémiologie génomique :
- Existe-t-il un génome de référence ?
- Existe-t-il un profil MLST ?
- Des modifications plus importantes sont-elles prévues ?
- Une contamination de l'ADN peut-elle se produire - métacommunauté attendue ?
- Existe-t-il déjà des gènes de résistance, de virulence, de mycotoxine spécifiques à la souche à analyser ?
Laissez-nous vous guider - de la conception à l'analyse.
Exemples de projets utilisant l'analyse d'épidémiologie génomique :
- Déterminer la pathogénicité des isolats d'ADN bactérien par MLST
- Détecter les SNV et les InDels
- Vérifier l'épidémiologie moléculaire des souches putatives porteuses de la virulence, les facteurs de mycotoxicité et la résistance aux médicaments
Applications liées à l'analyse d'épidémiologie génomique :
- Métagénomique des amplicons/ Shotgun
- Le séquençage de novo
- Le re-séquençage des microorganismes
- Transcriptomiques Shotgun
Déroulement
Le graphique ci-dessous illustre le déroulement typique d'un projet d'analyse d'épidémiologie génomique. Veuillez noter que notre process organisé en modules vous offre diverses options d'entrée et de sortie. Vous pouvez externaliser tout votre projet NGS ou seulement une partie de celui-ci à Microsynth.
Résultats
Généralement, dans les études d'épidémiologie génomique, une souche bactérienne est séquencée et analysée de diverses manières pour déterminer sa pathogénicité. Notre module d'analyse d'épidémiologie génomique vous aide à répondre, par exemple, aux questions suivantes :
- Comment la souche séquencée est-elle reliée à l'arbre phylogénétique déjà connu du microbe analysé ? (voir tableau 1)
- Quels sont les gènes de résistance, de virulence et de mycotoxine présents dans l'échantillon analysé ?
- Y a-t-il des variations telles que les variations d'un seul nucléotide (SNV) ou de petites insertions et délétions (InDels) par rapport à un génome ou des gènes de référence et quelle est leur conséquence ? (voir tableau 2)
- Quelle est la composition, si elle est présente, de la méta-communauté ? (voir tableau 3)
- Y a-t-il de nouveaux gènes qui présentent une homologie significative avec des familles de protéines connues ? (voir tableau 4)
Tableau 1. Résultat du Multi Locus Sequence Typing (MLST) montrant le type de séquence des espèces trouvées dans l'échantillon. Dans ce cas, le schéma utilisé pour le typage comprenait 7 gènes (arcC, aroE, glpF, gmk, pta, tpi et yqiL) pour identifier le type de séquence (ST) et le complexe clonal respectifs.

Tableau 2. Tableau récapitulatif du nombre de SNV et de petits InDels observés dans l'échantillon analysé, y compris le type de mutation (silencieuse et non silencieuse).

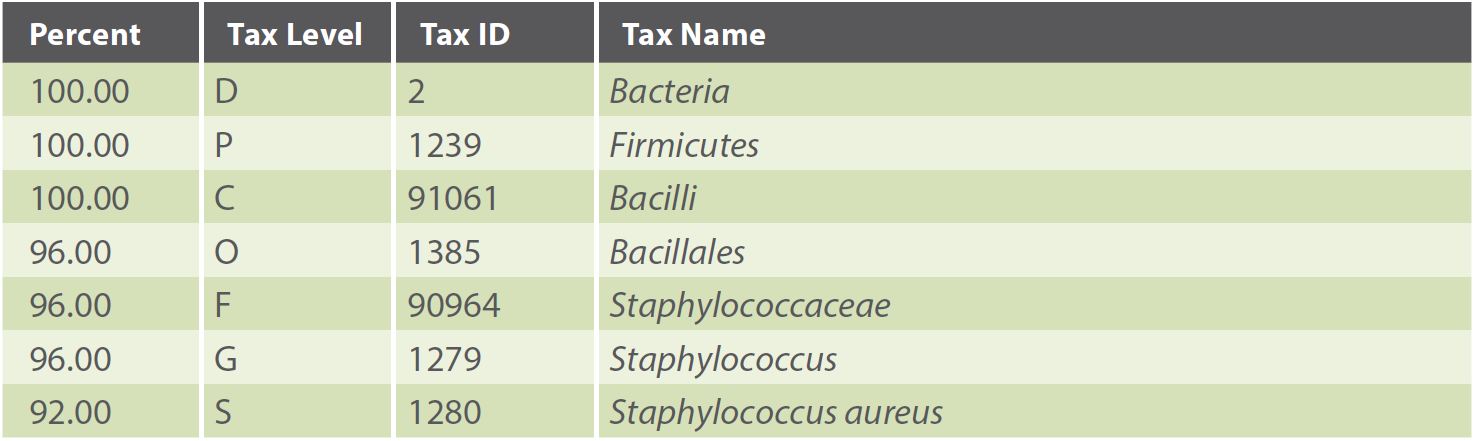
Tableau 3. Une partie du tableau de résultats d'une affiliation taxonomique par métagénomique Shotgun, montrant la composition de la communauté bactérienne trouvée dans l'échantillon analysé. Dans cet exemple, l'échantillon contient 96 % du genre Staphylococcus (niveau taxonomique : G) et 92 % sont identifiés comme des espèces de Staphylococcus aureus (niveau taxonomique : S).
Délai d'exécution
- Livraison des données dans les 20 jours ouvrables suivant la réception de l'échantillon (y compris la préparation de la librairie et le séquençage)
- 5 jours ouvrables supplémentaires pour l'analyse des données (bioinformatique)
- Service express possible sur demande



